terça-feira, dezembro 27, 2005
Mais três genomas
Desde que a sequência do genoma de Saccharomyces cerevisiae foi publicada (primeiro eucarionte com o genoma sequenciado) em 1996, os projectos de sequenciação de outros organismos não têm parado de aumentar, culminando na sequenciação dos tão aguardados, numa perspectiva antropocêntrica, Homo sapiens e Pan troglodytes. De acordo com o European Bioinformatics Institute, há 24 arquebactérias sequenciadas, 169 eubactérias, 33 eucariontes (mais 3 a partir de agora), 28 vírus e mais viróides, plasmídeos e DNA de mitocôndrias e cloroplastos.
A informação disponível permite fazer comparações filogenéticas entre seres vivos e é uma ferramenta de trabalho inestimável. Um exemplo disso é o da levedura S. cerevisiae. A base de dados do genoma desta levedura, Saccharomyces Genome Database, é duma facilidade de utilização verdadeiramente fantástica. As actualizações são frequentes com contributos da "yeast community" e já lá estão há muito tempo os dados obtidos com abordagens a nível global de genomas, proteomas e transcriptomas como os "microarrays" (aí está um exemplo do que a informação da sequência de genomas permite fazer).
quarta-feira, dezembro 21, 2005
Wiki científica

Via Nature, uma wiki científica, OpenWetWare, criada por cientistas de Harvard e do Massachusetts Institute of Technology destinada à partilha de protocolos e de dados entre grupos de investigação em Biologia. É uma novidade a acrescentar aos blogues de discussão de resultados de investigação científica antes da publicação dos artigos, ou após a respectiva publicação (a que já me referi anteriormente).
No mesmo artigo da Nature, referência à resistência por parte da comunidade científica à utilização de blogues: dos 20 milhões de blogues existentes, apenas algumas dezenas são blogues científicos.
terça-feira, dezembro 20, 2005
Clonagem de humanos

Em defesa da clonagem terapêutica e reprodutiva de humanos uma série de argumentos sólidos por parte de John Harris, professor de Bioética da Universidade de Manchester no seu recente livro "On Cloning" (Routledge). No caso da clonagem terapêutica, também sempre o considerei, afirma mesmo que é uma obrigação por parte da sociedade, tendo em conta os potenciais benefícios. Mesmo os argumentos que eu considerava irrebatíveis contra a clonagem reprodutiva aparecem aqui convincentemente refutados como o da variabilidade genética, da instrumentalização e da segurança. Este é, aliás, o único argumento que Harris considera decente (aos outros considera-os como retórica vazia e irracionais) na recusa da clonagem humana; basta pensar que a ovelha Dolly foi um caso de sucesso em 277 tentativas. De qualquer modo, refere que o princípio abstracto da segurança não é válido uma vez que a reprodução sexual e a reprodução medicamente assistida também não são isentos de problemas.
De leitura obrigatória para esclarecer muitas mentes.
sábado, dezembro 17, 2005
Um blogue por artigo científico
A blogosfera científica está a mover-se! Muitos investigadores usam blogues como locais de discussão em linha de resultados científicos. É muito interessante esta abertura à discussão em locais de acesso público de resultados de investigação. Será interessante seguir este processo para verificar como se conseguirá gerir a confidencialidade de resultados promissores com a exposição numa discussão aberta em linha e não em encontros científicos que contam como publicações oficiais.
É curioso também o recurso ao blogging por parte de cientistas de empresas farmacêuticas e a convivência, sem conflitos de maior, com as políticas de divulgação de resultados por parte das administrações. É claro que aqui o bom senso parece prevalecer, uma vez que, pelo menos nos casos relatados, houve uma definição clara dos limites de actuação tanto por parte dos bloggers como das administrações.
Tudo isto cheira a coisas novas que poderão surgir na comunicação de ciência!
domingo, dezembro 11, 2005
Arqueologia genética
quarta-feira, novembro 23, 2005
And now, something completely different!
segunda-feira, novembro 21, 2005
Bioinformática em expansão
De acordo com uma análise mencionada na Scientific Computing World de Outubro/Novembro, o mercado europeu de bioinformática vai sofrer uma expansão de cerca de 310 milhões de dólares para 710 milhões em 2011. A aposta na área da biologia computacional está patente no investimento privado (principalmente indústria farmacêutica na pesquisa de novos fármacos) mas também de institutos académicos sem fins lucrativos como Instituto Europeu de Bioinformática (http://www.ebi.ac.uk/). Cá em Portugal, foi criado o programa de doutoramento em Biologia Computacional (http://bc.igc.gulbenkian.pt/pdbc/) pela Fundação para a Ciência e Tecnologia, a Siemens SA Portugal e a Fundação Calouste Gulbenkian (para os alunos da Engenharia Biomédica esta pode ser uma oportunidade a considerar depois da graduação).
Interessante é esta área de criação recente necessitar de quadros com curricula "não convencionais", aliando Química-física à Biologia e estas à informática.terça-feira, novembro 08, 2005
Como é que uma proteína “enrola"?
Dada uma sequência de aminoácidos, pode-se prever a estrutura tridimensional da proteína? A resposta é não, excepto para algumas proteínas muito pequenas como a ubiquitina (76 aminoácidos) que foram “enroladas” graças ao projecto Rosetta@home (http://boinc.bakerlab.org/rosetta/) em que entusiastas de todo o mundo disponibilizam capacidade de processamento dos seus computadores pessoais para “enrolar” uma proteína (tal e qual como o projecto SETI@home de pesquisa de vida inteligente extraterrestre - já agora, http://setiathome.ssl.berkeley.edu/). Quando se aplicam os algoritmos existentes a proteínas maiores, a quantidade de possibilidades de estruturas terciárias é tão grande que a determinação da estrutura de menor energia tem sido condenada ao insucesso. Para diminuir o tempo necessário para os cálculos, Baker e colaboradores da Universidade de Washington (Bradley et al., 2005. Science 309: 1868-1871), desenvolveram uma metodologia que consiste em prever interacções de baixa energia em pequenas sequências de aminoácidos homólogas e mapear estas regiões na proteína em análise. Por outro lado, Ranganathan e colaboradores da Universidade de Texas Southwestern Medical Center (Socolich et al., Nature. 2005. 437: 512-518), usando análise estatística a alinhamentos múltiplos de sequência de uma família de proteínas, conseguiram desenhar proteínas artificiais que adquirem a estrutura tridimensional funcional típica desta família proteica. Seria interessante que estas duas abordagens opostas convergissem num modelo matemático comum de previsão da estrutura tridimensional de proteínas. Aposto que seria de uma robustez a toda a prova.
domingo, novembro 06, 2005
O genoma do chimpanzé

Com a sequenciação do genoma do chimpanzé (Mikkelsen et al., Nature 437: 69-87) é possível comparar os genomas do Homo sapiens com o de Pan troglodytes para tentar determinar que genes determinam as características humanas como a capacidade craniana, desenvolvimento do cérebro e bipedalismo.
As diferenças encontradas no tipo de mutações de um só nucleótido (que reflectem pressão selectiva) entre os dois genomas não são muito diferentes das encontradas entre genomas de humanos (apenas 1,23%). Aparentemente, a evolução para o ser humano deveu-se a uma proporção baixa de mutações vantajosas, ao contrário do que se poderia esperar.
Todavia, a questão poderá estar nas duplicações e inserções/eliminações de segmentos de cromossomas. As diferenças encontradas a este nível já são maiores (2,7% e 3%, respectivamente) do que as de mutações por substituição de um só nucleótido. Tomando apenas estes dados da quantificação de processos moleculares (sem atender às respectivas consequências evolutivas, até porque ainda são muito difíceis de prever), parece que a humanização poderá estar nas inserções/eliminações e duplicações. De acordo com Wen-Hsuing e Saunders do Department of Ecology and Evolution, University of Chicago (Wen-Hsiung and Saunders, Nature 437: 50-51), há cerca de 7000 elementos Alu (sequências de DNA móveis denominadas transposões que podem inserir-se noutra região do genoma) no genoma humano e apenas cerca de 2300 no chimpanzé. Já no que diz respeito a duplicações de segmentos maiores que 20 milhões de nucleótidos, 33% das duplicações "humanas" correspondem a regiões específicas de humanos (levando geralmente a aumento de expressão), enquanto que, no caso dos chimpanzés, cerca de 17% é que são de sequências específicas de chimpanzés.
Em suma, há 35 milhões de diferenças a nível de nucleótidos, 5 milhões a nível de inserções/eliminações e muitos rearranjos cromossomais. A chave da humanização deverá ser encontrada aqui. Para compreender como é que estas alterações no genoma se manifestam (agora sim, tentar entender as consequências evolutivas de difícil previsão), há, de acordo ainda com Wen-Hsiung and Saunders, três hipóteses:
1. Evolução de proteínas. Seria de esperar que genes codificando proteínas envolvidas na actividade cerebral tivessem, em humanos, mais mutações pontuais (substituição de um só nucleótido) que signifiquem alteração de aminoácidos (não-sinónimas) do que aquelas que não provoquem alteração no aminoácido que é codificado pelo codão (sinónimas). Na verdade, os genes que apresentam mais mutações não-sinónimas que sinónimas, codificam para proteínas envolvidas na interacção hospedeiro-patogénio, imunidade e reprodução. Aparentemente, não será por aqui que se poderá explicar a "humanização", se bem que esta análise seja fortemente influenciada pela tendência dos genes codificantes de proteínas do sistema imunológico e envolvidas na reprodução serem muito variáveis, pelo que a respectiva comparação com as outras proteínas, em termos de variabilidade, poderá não ser a mais correcta.
2. "Menos é mais". A evolução para humanos pode ter seguido esta via, ou seja, a perda de função por substituições não-sinónimas e inserções/eliminações (ou até perda física por eliminação) de alguns genes pode ter levado à "humanização". Como exemplos, temos a perda de pilosidade corporal e a manutenção de características infantis no adulto como o crânio de elevadas dimensões em relação ao corpo (designado por neotenia, e que está ligada à curta gestação dos seres humanos, se comparada com outros seres vivos, devido às grandes dimensões do crânio).
3. Alterações da regulação da expressão. A regulação da expressão de genes está ainda mal avaliada pois é necessário identificar as regiões dos genomas que têm essa função, para além dos promotores de cada gene que podem ser afectados por substituições e inserções/eliminações. É uma hipótese promissora se considerarmos os exemplos da intensa actividade de regulação da expressão génica que permite que células de um mesmo organismo (logo com a mesma informação genética) possam ter morfologia e funções metabólicas totalmente diferentes. Este terceiro mecanismo é defendido por Hill e Walsh (Nature 437: 64-67), que no artigo no mesmo número da Nature, referem haver indicações de vários estudos apontando para mais modficações da expressão de genes no cérebro ao longo da história evolutiva da linha humana em relação à dos chimpanzés. Referem ainda outros estudos que apontam para mais modificações da expressão de genes no cérebro do que no fígado na evolução humana.
As diferenças evolutivas fundamentais que nos separam de todos os outros seres vivos parecem corresponder a alterações genéticas ainda não compreendidas mas que, já o sabemos, não são aquelas evidências simples e praticamente auto-explicativas. Os genomas são complexos e os seres vivos ainda o são mais; pequenas alterações podem ter grandes consequências e grandes alterações nenhuma.
terça-feira, outubro 18, 2005
Congresso Micro'05-Biotec'05

Para os meus alunos das Engenharias Biológica e Biomédica, fica a sugestão do congresso Micro'05-Biotec'05 a decorrer na Póvoa de Varzim, de 30/11 a 3/12 (http://www.microbiotec2005.info/), da Sociedade Portuguesa de Microbiologia e da Sociedade Portuguesa de Biotecnologia. A organização está a cargo, em conjunto, do Departamento de Biologia da Universidade do Minho e da Escola Superior de Biotecnologia da Universidade Católica Portuguesa. Já é um sucesso de participação: 462 comunicações submetidas.
segunda-feira, outubro 17, 2005
Tarefa de paciência

Eis uma aplicação prática da genética de grande impacto social: a identificação de indivíduos desaparecidos devido a conflitos militares, catástrofes naturais ou soldados participantes em batalhas (ver Público de 15/10/05, página 30). É impressionante a quantidade de trabalho envolvido na identificação de vítimas de catástrofes ou de conflitos militares, pois não basta fazer a colheita de material biológico para fazer a análise (o que já é difícil em ambiente caótico pós-catástrofe), muitas vezes com material já em degradação. É preciso confrontar os perfis genéticos com os de familiares de desaparecidos e também cruzar as informações genéticas com outras de carácter antropológico e ainda outras, diversas, como por exemplo a presença de objectos pessoais junto ao cadáver. É um trabalho longo e lento que requer paciência como refere Thomas Parsons, consultor da Comissão Internacional para Pessoas Desaparecidas (ICMP), pois em muitos casos, os conflitos ainda não estão completamente resolvidos e é preciso envolver familiares em populações envolvidas em conflitos étnicos como por exemplo na Bósnia e Sérvia. Do conflito na ex-Jugoslávia, a ICMP tem o perfil genético de 15000 ossos, o que possibilitou a identificação de 8000 vítimas. Citando Thomas Parsons nesta entrevista: "...à medida que são identificadas mais pessoas, mais consciência há de que isto só é possível através da cooperação entre pessoas que já não estão em guerra. Serve para sarar as feridas da sociedade e permite a consciencialização de que aconteceu a todos uma tragédia e há uma solução comum."
segunda-feira, outubro 10, 2005
Vírus "ressuscitado"
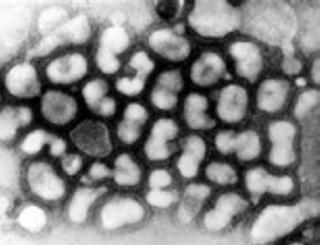
A palavra ressuscitado é demasiado forte, pois implica regressar à vida e isto é coisa que não se aplica aos vírus. Mas não deixei de pensar nisso ao ler este artigo (Tumpey et al., 2005. Science 310: 77-80). Neste trabalho, os autores recuperaram RNA genómico do vírus da gripe que provocou a pandemia de 1918 a partir pulmões preservados em formalina de autópsias de pacientes e de pulmões de cadáveres que estavam enterrados no Alaska em solo gelado. Após sequenciação do seu genoma, determinaram a existência de 8 sequências genómicas codificantes tal como, obviamente, os seus "parentes" Influenza actuais.
Por genética reversa criaram, a partir duma estirpe viral actual, uma estirpe portadora das sequências genómicas da estirpe da pandemia de 1918 (mais uma vez reforço as aspas do ressuscitado do título uma vez que a recriação do vírus não é perfeita pois faltam as sequências das extremidades 3' e 5' do seu genoma que, não sendo codificantes, poderão regular a expressão dos genes virais e, assim, influenciar a replicação viral). Criaram em simultâneo outras estirpes recombinantes, contendo um ou mais dos genes da estirpe de 1918. Em laboratório, utilizando hospedeiros como células de rim de cão e ovos de galinha com embrião, testaram a replicação destas estirpes. O modelo que usaram para testar a patogenicidade foi o ratinho, e verificaram que a estirpe criada é muito mais virulenta que as actuais (até 39000 vezes). Por comparação dos dados de patogenicidade e replicação, concluiram que os genes codificantes para uma proteína do envelope viral (hemaglutinina) e os codificantes para a RNA polimerase são essenciais para a virulência.
Noutro trabalho (Taubenberger et al., 2005. Nature 437: 889-893), foram analisadas as sequências da polimerase do vírus de 1918 (também estudadas no trabalho de Tumpey e colaboradores) e comparadas com as de aves. A elevada homologia sugeriu que estas sequências virais têm origem em hospedeiros de aves e que a sua capacidade de transmissão a humanos se deveu a esta adaptação do vírus e não à recombinação entre estirpes virais humana e de ave coexistindo na mesma célula hospedeira humana. Identificaram mudanças de aminoácidos na polimerase por comparação com estirpes actuais que são semelhantes às apresentadas pela estirpe H5N1 responsável pela actual gripe das aves que já provocou a morte a cerca de 60 pessoas na Ásia. Ou seja, parece que esta estirpe da gripe das aves está a seguir o caminho evolutivo que leva à capacidade de transmissão a humanos com elevada virulência.
As vacinas vêm sempre com atraso uma vez que só se podem preparar a partir das estirpes existentes. Como de um ano para outro há acumulação de novas mutações, não se consegue imunização contra as estirpes que surgirão no próximo período de incidência da gripe. Esta "arqueologia biológica" realizada nestes trabalhos, permitiu a descoberta de sequências genómicas que poderão estar envolvidas na virulência do vírus (codificantes para a hemaglutinina e para a polimerase) e na mudança de hospedeiro de aves para humanos (codificantes para a polimerase com origem no hospedeiro avícola). Se é este o fenómeno que faz despoletar o aumento de virulência do vírus, incluindo a capacidade de transmissão a humanos, então deu-se um passo enorme no combate a estas pandemias de gripe, nomeadamente na possibilidade de criação de vacinas contra estirpes altamente virulentas.
Uma questão interessante se levanta nesta investigação. Será sensato publicar estas sequências com potencial de aproveitamento bélico, nomeadamente para grupos terroristas? E ao não publicar, não haverá o risco de dificultar a livre investigação e atrasar o combate a uma pandemia que os peritos dizer estar iminente? No entender do corpo editorial da revista Science, a melhor decisão é a da publicação. Sustentam esta posição na impossibilidade de previsão do efeito de observações científicas no estímulo de criação de novos tratamentos para o controlo de pandemias. Ao não restringir a divulgação de observações científicas, contribui-se genericamente para o surgimento de novas descobertas com aplicação útil sendo, eventualmente, maior o benefício para a sociedade do que o risco de potencial aplicação bélica. Esta será, na minha opinião também, a melhor maneira de contribuir para a prevenção da pandemia.
(fotografia: Centers for Disease Control and Prevention, USA)
quinta-feira, outubro 06, 2005
Incrível forma de vida

Se não tivesse sido publicado na Science (La Scola, B. et al. 2003. Science, 299. 2033; Raoult, D. et al., 2004. Science, doi: 10.1126/science.1101485; ambos disponíveis online), eu duvidaria da veracidade deste ser. Após a sequenciação do seu genoma pelo grupo de Jean-Michel Claverie, foi possível determinar que o mimivirus possui características tão extraordinárias para um vírus que parece apenas produto da imaginação. É o tamanho (enorme, comparado com outros vírus e maior que o de algumas bactérias), a composição em ácidos nucleicos (DNA e RNA, ao contrário dos vírus "normais"), o tamanho do genoma (1260 genes, comparável a muitas bactérias), a capacidade de produzir proteínas pelos seus próprios meios e a capacidade de reparação do seu próprio genoma, entre outras.
Se para os vírus (excluindo este, claro), não haverá grandes problemas em não os classificar como formas de vida (a imagem de cristais de partículas virais num tubo de ensaio é particularmente elucidativa), quando olhamos para este mimivirus, confrontamo-nos com a dificuldade em colocar esta "coisa" num dos lados da fronteira que delimita os seres vivos. Aquela ideia de simplicidade dos vírus constituídos por uma carapaça proteica envolvendo o genoma viral (composto por DNA ou RNA, e não ambos), codificando um reduzido número de proteínas e dependendo em larga escala da maquinaria enzimática da replicação, transcrição e tradução, está, assim, posta em causa.